
发表期刊:Nature Communications
影响因子:12.121
研究方法:m6A- meRIP-seq、meRIP-qPCR等
文章链接:RNA m6A methylation orchestrates cancer growth and metastasis via macrophage reprogramming
研究内容
(1)骨髓特异性的Mettl3缺失促进肿瘤生长和转移。
为了研究m6A修饰在巨噬细胞中的作用,作者将WT BMDMs(骨髓源性巨噬细胞)与B16细胞共孵育,发现Mettl3的表达明显减少。为了研究骨髓细胞中的Mettl3是否能够调节肿瘤的进程。作者将Mettl3fl/fl小鼠与Lyz-cre小鼠杂交敲除了髓腔室中的Mettl3。然后,对Mettl3fl/flLyz2+/+ (WT)和Mettl3fl/flLyz2cre/+ (KO)小鼠的BMDMs和腹腔巨噬细胞(PMs)进行qRT-PCR和western blotting检测。结果表明,来自KO小鼠的BMDMs和BM中Mettl3 mRNA表达分别下降了约75%和80%。此外,KO BMDMs中mRNA上m6A修饰水平低于WT。作者将B16或LLC细胞皮下注射到WT或KO小鼠中,KO小鼠的肿瘤生长速度明显快于WT小鼠,这些敲除Mettl3小鼠的生存时间缩短(图1a-d)。通过尾静脉将B16或LLC细胞注射到小鼠体内,生物发光成像显示,与WT小鼠相比,KO小鼠的肺转移增强(图1e, f)。组织学检查显示,与WT小鼠相比,KO小鼠有更大、更多的肺转移结节且Ki67染色增加(图1g-j),导致KO小鼠比WT小鼠总生存时间缩短(图1k)。此外,与正常组织中的巨噬细胞相比,TAMs中Mettl3的表达减弱(图1 l)。上述结果说明Mettl3在骨髓细胞中的缺失会导致肿瘤的生长和增殖。
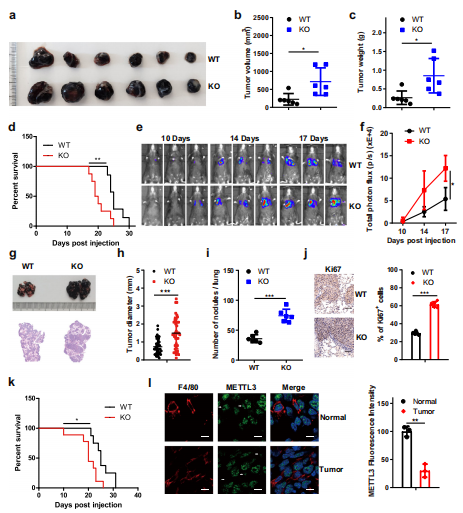
为了探讨BMDMs中Mettl3的缺失是否会促进肿瘤生长,作者建立了巨噬细胞和肿瘤细胞的混合模型。用CFSE标记BMDMs,并将B16或LLC细胞共注入小鼠皮下F4/80免疫荧光染色结果显示CFSE标记的细胞共注射BMDMs可在肿瘤中存活2周。进一步的分析表明,B16或LLC细胞与KO BMDMs共注射可加速肿瘤的生长(图2a-f)。接下来,在实验前两天通过静脉注射氯膦酸脂质体来减少小鼠的巨噬细胞,然后将B16细胞静脉注射到小鼠体内,生物发光成像显示,巨噬细胞耗竭消除了WT和KO小鼠肿瘤生长和转移的差异(图2g, h),并通过肺肿瘤组织学分析证实了这一点(图2i, j)。消耗了巨噬细胞的小鼠的生存期得到延长,用氯膦酸脂质体处理的WT和KO小鼠之间没有明显差异(图2k)。综上所述,巨噬细胞中METTL3在促进肿瘤进展中发挥重要作用。
(2)巨噬细胞中Mettl3的缺失通过增强M1-和M2-like TAM和Treg对肿瘤的浸润来重塑肿瘤微环境。
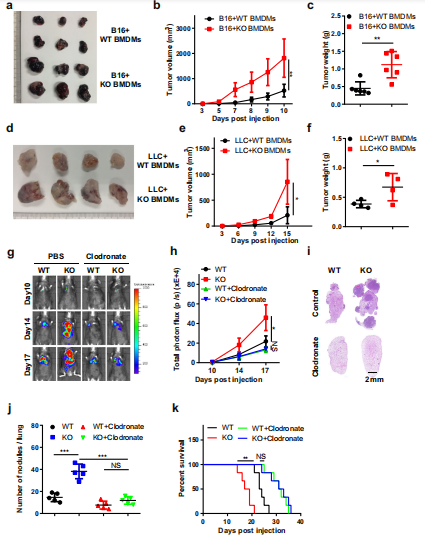
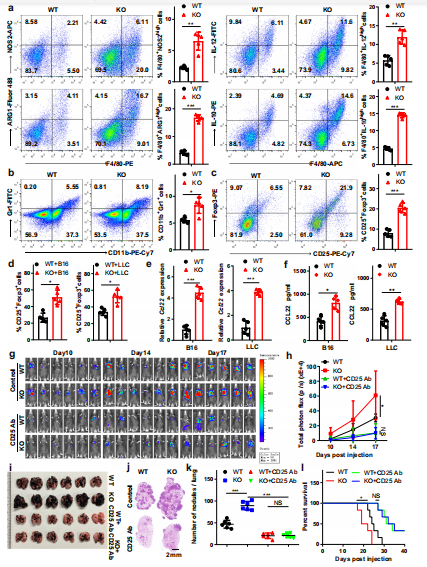
为了评估Mettl3对免疫微环境的影响,采用流式细胞术对脾和肺进行免疫表型分型,发现KO小鼠的M1巨噬细胞, M2巨噬细胞和调节性T细胞的总体百分比增加。为了进一步研究Mettl3对肿瘤微环境的影响,通过单细胞RNA测序分析了WT或KO小鼠肿瘤中的CD45+细胞。KO小鼠的肿瘤与WT小鼠的肿瘤在免疫细胞的组成上存在差异,包括TAMs、MDSCs和CD4+的数量增加,T细胞以及粒细胞和CD8+ T细胞数量减少的趋势。此外,通过流式细胞术对含有mettl3-缺陷巨噬细胞的肿瘤组织进行免疫表型分析,发现M1-和M2-like TAMs总体百分比增加(图3a)。此外,MDSCs和调节性T细胞的数量有增加的趋势(图3b-d)。先前的研究表明,Treg在各种肿瘤组织中的迁移和浸润似乎依赖于肿瘤细胞或浸润巨噬细胞产生的CCR4配体(CCL22)的表达。通过qRT-PCR和ELISA检测发现,在mettl3缺失的BMDMs和TAMs中,CCL22表达明显上调(图3e, f)。作者研究了注射抗CD25抗体对Treg水平的影响,发现Treg水平有75%的下降。随着Treg细胞数量的减少,Th1细胞和IFN-γ+ CD8 T细胞在肿瘤细胞中增加,而Th2细胞基本未受影响。进一步分析表明WT和接受抗CD25抗Treg消除抗体的KO小鼠在肿瘤生长或转移方面没有明显差异,但与WT小鼠相比,在未处理组,KO小鼠的肿瘤生长增加(图3g-k)。去掉Treg后,WT和KO小鼠的存活率差异也消失了(图3l)。这些数据表明髓系细胞中Mettl3的缺失促进了肿瘤的生长和转移,其方式依赖于M1/M2-like TAM浸润和Treg募集。
(3)Mettl3敲除通过NF-kB / STAT3轴促进BMDMs中M1和M2极化。
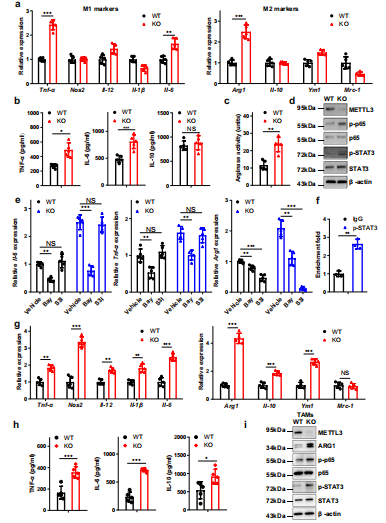
为了研究Mettl3在巨噬细胞极化中的潜在作用,使用LPS和IFN-γ诱导M1巨噬细胞或IL-4诱导M2巨噬细胞。qRT-PCR检测了M1和M2巨噬细胞相关基因的表达,发现在mettl3缺失的BMDMs中TNF-α、IL-6和Arg1的表达明显上调(图4a)。ELISA还发现Mettl3缺失的BMDMs中TNF-α和IL-6表达上调,IL-10表达相对不变(图4 b)。在Mettl3缺失的BMDMs中也观察到精氨酸酶活性的增加(图4c)。在BMDMs中评估了p65,观察到p65和STAT3磷酸化水平在mettl3缺失的巨噬细胞中升高(图4d)。NF-κB的抑制降低了TNF-α、IL-6和Arg1,而抑制STAT3通路降低了WT和KO BMDMs中Arg1的表达(图4 e)。芯片分析表明p-STAT3特异性结合到Arg1的启动子上(图4f)。接下来从携带肿瘤的小鼠中收获并用流式细胞术检测TAMs结果显示,KO TAMs中M1和M2巨噬细胞相关基因表达同时增加,这与p65和STAT3磷酸化和ARG1表达增强有关(图4g-i)。综上所述,这些数据表明Mettl3的缺失导致NF-κB通路和STAT3信号通路的激活,从而导致M1和M2-like巨噬细胞极化。
(4)肿瘤细胞增强细胞因子的产生,并在mettl3敲降巨噬细胞中激活NF-κB和STAT3。
由于NF-κB和STAT3可受细胞外源性因子TNF-α或IL-6刺激BMDMs调节。免疫印迹结果显示,KO BMDMs诱导p-p65和p-STAT3的能力比WT更明显。qRT-PCR分析显示TNF-α处理后TNF-α和IL-6的表达增加,通过bay11-7082阻断NF-κB通路后逆转,而IL-6作用后,Arg1的表达增加,通过STAT3抑制剂S3I-201处理后逆转。这些结果提示,NF-κB/IL-6/STAT3信号轴在KO BMDMs中比WT BMDMs更活跃。
为了评估NF-κB/IL-6/STAT3信号轴在肿瘤微环境中的作用,将B16或LLC细胞与BMDMs孵育,发现IL-6在B16和LLC细胞中表达上调,p65磷酸化水平增加,并通过bay11 -7082阻断NF-κB通路后逆转(图5a)。进一步发现TNF-α治疗不仅增加了p65的磷酸化也增加IL-6的表达,用NF -κB抑制剂处理可逆转肿瘤细胞中IL-6的表达(图5b)。此外,WT或KO BMDMs用bay11 -7082或抗TNF-α抗体前处理然后与B16或LLC细胞共培养。qRT-PCR分析显示,bbay11 -7082或抗TNF-α抗体的加入抑制了IL-6的表达(图5c),伴随p65和STAT3磷酸化降低(图5 d)。此外,在mettl3缺失的BMDMs与B16或LLC细胞共培养时,与WT BMDMs相比,Arg1的表达明显降低(图5e, f)。总之,这些数据表明,mettl3缺失的巨噬细胞通过调节细胞因子反应重塑了肿瘤微环境。
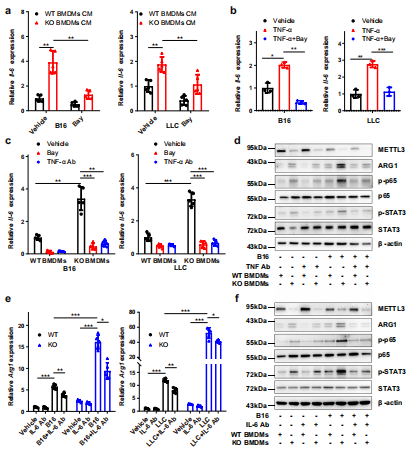
(5)METTL3的缺失会损害YTHDF1介导的SPRED2翻译。
探讨m6A在巨噬细胞重编程中的作用,对WT和KO BMDMs进行了m6A meRIP-seq,生信分析得到了m6A峰密度图(图6a),Motif分析发现了m6A修饰的保守Motif“GGAC”(图6b)。为确定m6a调控的巨噬细胞极化潜在的目标基因,作者在MAPK通路相关基因和70个m6A下调基因中取交集获得候选基因。在这些基因中,Spred2上m6A水平明显降低(图6c, d)。此外,meRIP-qPCR 证实Spred2是m6A调控的靶基因(图6e)。随后在KO BMDMs和TAMs中发现,SPRED2蛋白表达降低,而mRNA水平没有明显改变(图6f)。mRNA衰减实验表明,m6A修饰对Spred2的衰减没有明显影响(图6g)。因此推测在Mettl3缺失的BMDMs中,SPRED2蛋白表达的下调可能是由于YTHDF1控制的蛋白质翻译效率的差异所致。
然后作者通过多聚体分析WT和KO BMDMs将RNA分成非翻译部分,翻译起始部分和翻译活性多聚体(图6h)。qRT-PCR结果显示,mettl3缺失的BMDMs翻译活性多聚体中Spred2 mRNA水平明显低于WT BMDMs(图6i)。然后检测了METTL3是否被招募到Spred2启动子中并影响Spred2的表达,结果表明野生型METTL3 和催化死亡突变体METTL3可影响荧光素酶活性。为了研究CDS中m6A甲基化是否能调控SPRED2的表达,将m6A修饰保守位点GGAC突变为GCTC (SPRED2 -CDS mut1或SPRED2 -CDS mut2)或同时突变(SPRED2 -CDS mut1/2)。其中Spred2-CDS mut2和Spred2-CDS mut1/2降低了SPRED2水平而Spred2-CDS mut1没有,这表明Spred2-CDS mut2中的m6A基序是表达调控的主要位点(图6j, k)。此外,RNA-IP结果显示,YTHDF1和Spred2在Mettl3 KO植株中互作下降。为了证实YTHDF1在m6a调控SPRED2表达中的作用,在BMDMs中下调了YTHDF1的表达。YTHDF1基因敲除明显减弱SPRED2在BMDMs中的表达,增加了ERK, NF-κB和STAT3磷酸化。qRT-PCR分析显示,YTHDF1敲除后Tnf-α、Il-6、Arg1和Ccl22的表达增加。此外,BMDMs中SPRED2的下调导致ERK、NF-κB和STAT3磷酸化上调,并伴有Tnf-α, Il-6, Arg1和Ccl22上调表达。总的来说,BMDMs中Mettl3的丢失导致BMDMs中ythdf1介导的SPRED2的翻译和ERK, NF-κB和STAT3磷酸化。
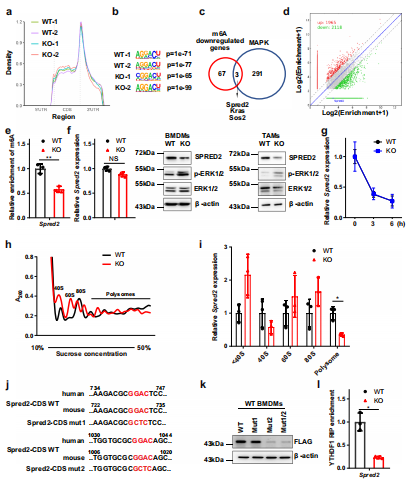
(6)髓系细胞中Mettl3的缺失会损害PD-1阻断治疗B16黑色素瘤的疗效。
为了评估靶向METTL3激活抗肿瘤反应的潜力,在B16肿瘤转移模型中测试了抗PD-1治疗。正如预期的那样发现在KO小鼠中,B16肿瘤对抗pd -1治疗的反应较低,这增加了肿瘤生长和肺转移,缩短了生存时间(图7a-f)。说明Mettl3缺乏的髓系细胞参与了B16肿瘤对抗PD-1治疗产生耐药性。
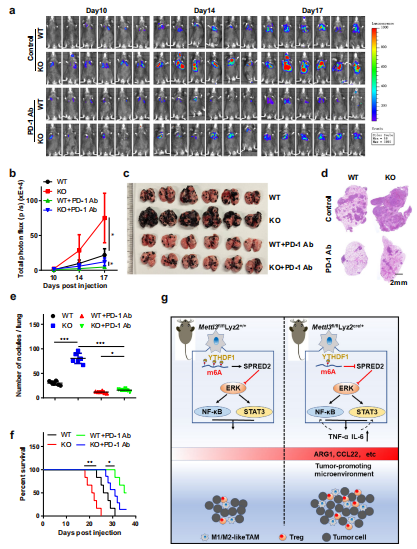
总结
这篇文章结合m6A meRIP-seq、 meRIP-qPCR等实验手段探究了甲基转移酶METTL3的髓样特异性缺失对小鼠肿瘤的生长和转移的影响,揭示了Mettl3确实可通过影响巨噬细胞重编程增强小鼠肿瘤模型中的肿瘤生长和转移,并减弱PD-1阻断疗法。该研究表明巨噬细胞中的m6A甲基化酶是潜在的癌症治疗靶标,为靶向治疗癌症提供了新的思路。
云序生物m6A修饰研究五大模块
01 m6A RNA修饰测序
m6A RNA修饰测序(m6A-seq)
对m6A RNA甲基化,目前流行的检测手段为m6A-Seq技术,适用于m6A RNA甲基化谱研究,快速筛选m6A RNA甲基化靶基因。云序可提供mRNA和多种非编码RNA的m6A测序:
- m6A 全转录组测序(涵盖mRNA,LncRNA,circRNA)
- m6A LncRNA测序(涵盖LncRNA和mRNA)
- m6A Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
- m6A mRNA测序
- m6A miRNA测序
LC-MS/MS检测整体RNA修饰水平
精准高效,可以实现一次检测,9类修饰水平检测,一步到位。
比色法
快速检测m6A整体甲基化水平
03 m6A RNA修饰上游酶的筛选
m6A RNA修饰相关酶PCR芯片
寻找上游直接调控m6A RNA甲基化的甲基转移酶。
04 m6A RNA修饰靶基因验证
meRIP-qPCR
云序提供各类不同修饰的meRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证RNA修饰靶基因表达水平。
05机制互作研究
5.1 RIP-seq/qPCR
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
5.2 RNA pull down -MS/WB
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
5.3 双荧光素酶实验
验证两基因互作,研究相应的分子调控机制。
云序生物服务优势
优势一:发表10分以上文章zui多的m6A RNA甲基化测序服务平台。云序已累计支持客户发表42篇高水平文章,合计影响因子300分+,是国内支持发文多、累计影响因子高的公司。
优势二:至今完成4000+例 m6A测序样本,覆盖医口、农口等各类样本。
优势三:检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:提供m6A一站式服务:m6A整体水平检测、m6A测序、MeRIP-qPCR验证、RIP和RNA pull-down。
优势五:率先研发超微量MeRIP测序,RNA量低至500ng起。
优势六:国内zui全的RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、O8G、ac4C乙酰化和2'-O-甲基化测序。
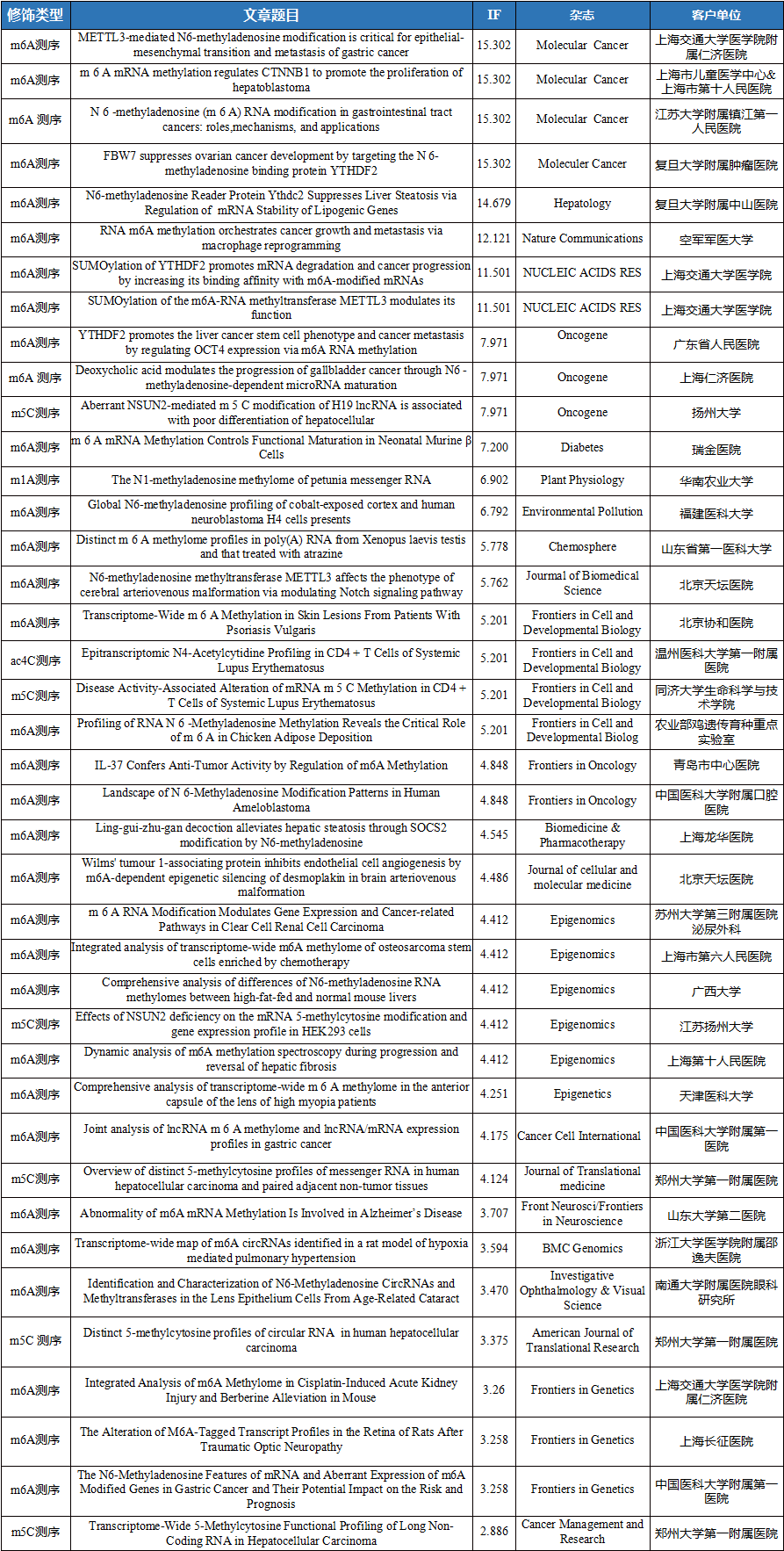
云序客户RNA修饰文章列表
相关产品
往期回顾
上海云序生物科技有限公司 商家主页
地 址: 上海市松江区莘砖公路518号24号楼4楼
联系人: 戴小姐
电 话: 021-64878766
传 真: 021-64878766
Email:market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
相关咨询
杨宝峰院士团队RNA修饰又一成果 | 云序ac4C acRIP-seq助力揭示心脏I/R损伤的作用机制 (2024-12-03T00:00 浏览数:12654)
杨宝峰院士团队最新成果 | 云序助力揭示RNA修饰m7G调控心肌肥厚的机制研究 (2024-11-13T00:00 浏览数:14443)
Nature子刊| 重磅综述!一文总结「m6A修饰非编码RNAs」在各类肿瘤中的调控机制及作用 (暂无发布时间 浏览数:11449)
研究速览-eccDNA 2023年最新进展大放送! (暂无发布时间 浏览数:12657)
云序生物MeRIP-qPCR技术干货 (暂无发布时间 浏览数:12588)
技术干货| “eccDNA碱基序列的获取及引物设计”方法教程 (暂无发布时间 浏览数:11760)
云序客户m6A高分文章|揭示组蛋白乙酰化与m6A修饰在眼部黑色素瘤发生中的共同作用机制 (暂无发布时间 浏览数:6467)
Nat Biotechnol IF=47 | BID-seq:一种基于单碱基分辨率的假尿嘧啶(Ψ)修饰定量测序检测方法 (暂无发布时间 浏览数:6374)
北大伊成器团队Nature Reviews重磅发文:非m6A热门修饰调控与功能一文速览! (暂无发布时间 浏览数:10027)
用户文章m6A专题|IF=9.8|m6A去甲基化酶ALKBH5缺乏会加重钴致神经退行性损伤 (暂无发布时间 浏览数:6337)
